
近日,北京基因组所(国家生物信息中心)原刘凡研究组与荷兰伊拉斯谟大学Manfred Kayser团队研发了一种能够集成分析多个全基因组关联研究的方法C-GWAS,并利用该方法分析了人类78个面部形态表型,发现了17个影响脸型的新遗传位点。相关研究成果发表于Nature Communications。

在方法设计层面,作者对多维GWAS统计量构成的相关性矩阵进行了分解,来区分由可解释遗传因素导致的 “效应相关性” 和由不可解释与非遗传因素导致的 “背景相关性”,进而依据效应和背景相关性相对强度优选合适的统计量进行集成分析,同时引入自适应迭代算法以甄别部分遗传变异仅对特定表型子集有效应的情况,从而实现对遗传多效性检出率的最大化。为了克服由于多重迭代优选引起的统计量膨胀,作者通过计算机模拟获得统计量在零假设下的真实分布,并利用其与均一分布的对应关系,对最终观测到的统计量进行校正,确保了C-GWAS结果和标准GWAS结果可直接在相同显著阈值下进行比较。通过大规模数据模拟发现,与多种其它方法相比,C-GWAS在不同复杂场景下的统计功效和稳定性均展示出明显提升。

不同统计量在效应和背景相关性相对强度变化时的统计效能变化趋势
人类面部形态代表了一组多维、可遗传且相互关联的复杂表型。作者应用C-GWAS集成分析了78个面部形态的GWAS,结果显示C-GWAS的检出率是传统方法的3倍,并发现了17个影响脸型的新遗传位点。
论文通讯作者、中科院北京基因组研究所研究员刘凡表示:“这显著提升了脸型的遗传解释度,并且挖掘出来的基因具有更明确的生物发育学功能,表明C-GWAS在解析多维复杂的表型遗传结构中具有很大优势。从现有结果看,C-GWAS是一种能够对多表型GWAS汇总数据进行集成分析的高效算法,其对遗传多效性高度敏感,并且在复杂场景下有很强的稳定性。”
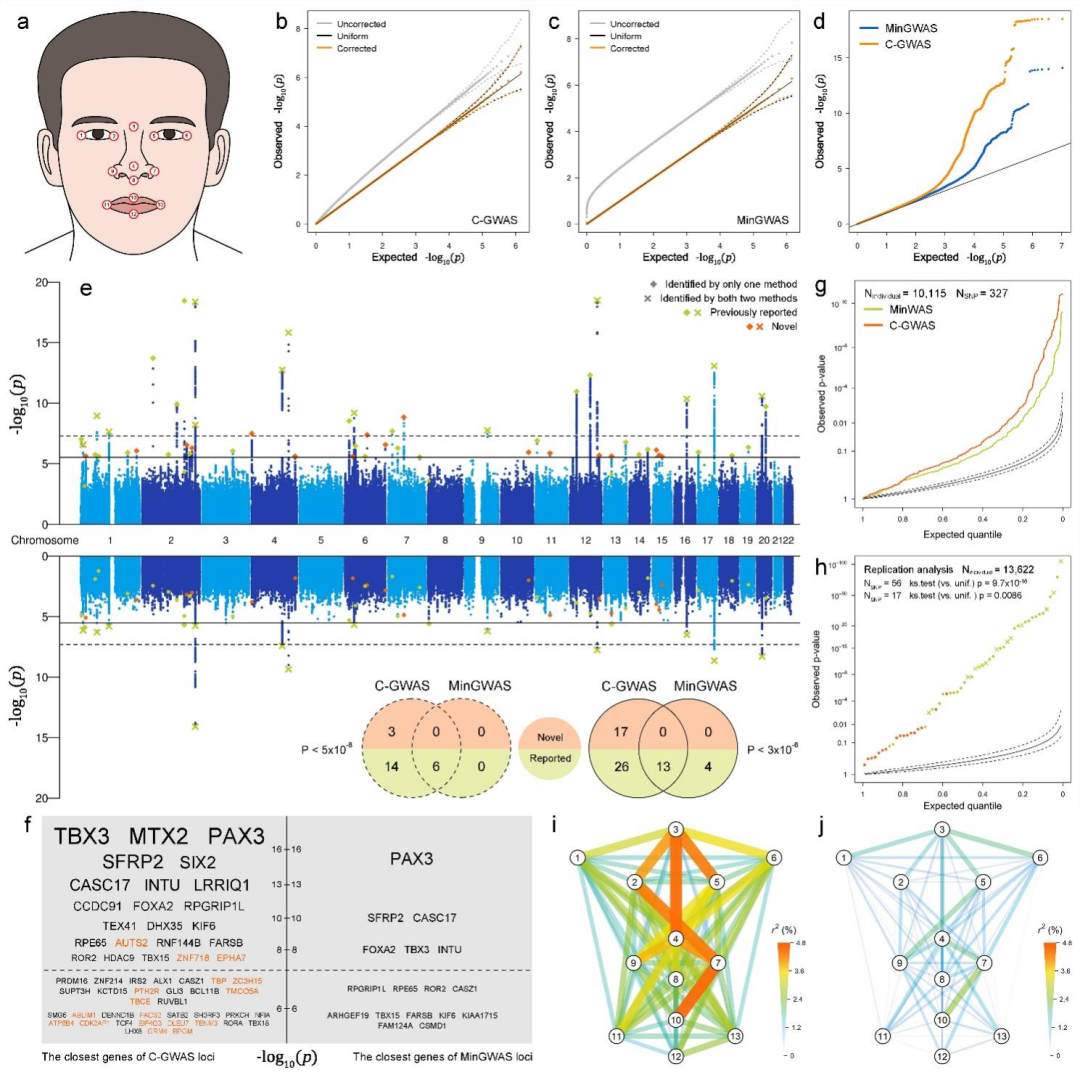
C-GWAS与标准GWAS方法应用于78个面部形态GWAS集成分析的结果比较
在新发现的17个影响面部形态的遗传位点中,有13个位点位于CNCC中活性调控元件附近,或在脑垂体等多个组织中表现出与表达数量性状位点(Expression quantitative trait loci, eQTL)信号的高度共定位。其中,与面部宽度和长度相关的CDK2AP1内含子中的多态性rs10773002,其附近的调控元件在CNCC中调控CDK2AP1的表达,且该位点在多组织中与CDK2AP1的eQTL高度共定位,CDK2AP1编码的蛋白在细胞周期、胚胎干细胞分化和表观遗传调控中发挥作用。这些证据提示该位点通过调控CDK2AP1的表达参与到面部形态形成的过程中。
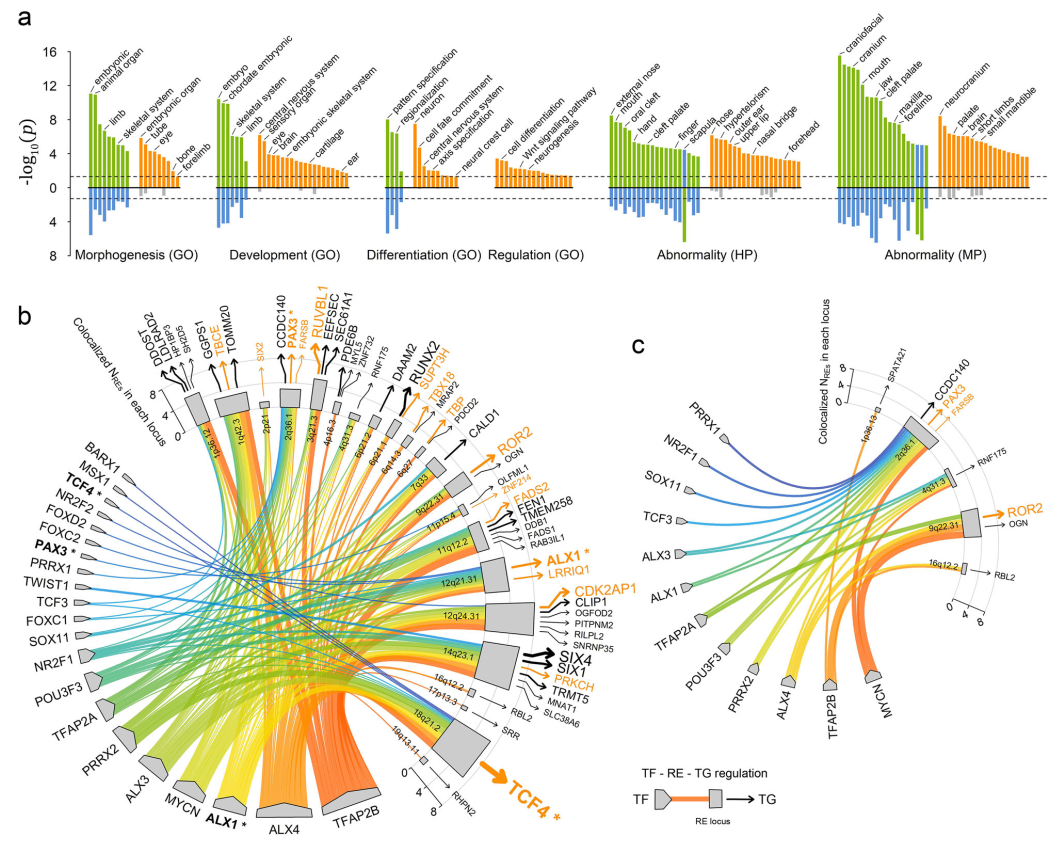
C-GWAS与标准GWAS确定的候选基因所富集到的生物通路与表型(a)和与CNCC中调控网络重叠情况(b,c)的比较
C-GWAS对人类面部形态表型的分析成功发现了一批新遗传位点和功能性基因,加深了人们对脸型遗传结构的理解,未来,C-GWAS将被用于解析更高维复杂表型的遗传结构,为人类表型组间的共享遗传因素网络的描绘提供技术支持。
参考资料:
www.nature.com/articles/s41467-022-35328-9
<END>













































 浙公网安备33011002015279
浙公网安备33011002015279 本网站未发布麻醉药品、精神药品、医疗用毒性药品、放射性药品、戒毒药品和医疗机构制剂的产品信息
本网站未发布麻醉药品、精神药品、医疗用毒性药品、放射性药品、戒毒药品和医疗机构制剂的产品信息
收藏
登录后参与评论
暂无评论